Wissenschaftler der Abteilung Biochemie am Mannheim Institute für Innate Immunoscience (MI3) der Medizinischen Fakultät Mannheim der Universität Heidelberg haben eine Methode entwickelt, mit der sie erstmals die Dauer der Beladungszeit neu synthetisierter Boten-RNA (mRNA) mit Ribosomen in der Zelle messen können. Johanna Schott aus dem Team von Georg Stoecklin machte dabei die überraschende Beobachtung, dass die Beladungszeit verschiedener RNA-Transkripte unterschiedlich lang und abhängig von der Halbwertszeit der mRNA ist. Die Beladung von mRNA mit Ribosomen erweist sich damit als ein wenig erforschter Mechanismus der Genregulation. Die Arbeit ist aktuell in Nature Methods publiziert*.
mRNAs sind Abschriften von aktiven Genen der Erbinformation DNA, die als Bauanleitung für die Proteinbiosynthese dienen. Die Transkripte werden im Zellkern synthetisiert und anschließend ins Zellplasma transportiert, wo sich mobile Proteinfabriken, die Ribosomen, an sie heften und den Bauplan in die Aminosäuresequenz des zu synthetisierenden Proteins übersetzen. Wie viel von jeder Art Protein hergestellt wird, hängt dabei nicht nur von der Anzahl der kodierenden mRNAs ab, sondern auch davon, wie effizient diese von Ribosomen abgelesen (translatiert) werden.
„Ursprünglich wollten wir vor allem wissen, wie lange es tatsächlich dauert, bis eine noch junge, gerade aus dem Zellkern exportierte mRNA ihre volle Produktivität erreicht hat“, berichtet die Leiterin der Studie, Johanna Schott. Als die Wissenschaftler die neue Methode entwickelt hatten, wurden sie jedoch von dem Ergebnis überrascht: „Einerseits brauchen die mRNAs im Schnitt wesentlich länger, um ihre volle Beladung mit Ribosomen zu erreichen, als bisher vermutet. Noch interessanter aber ist die Beobachtung, dass es zwischen verschiedenen mRNAs Unterschiede in der Dauer des Beladungsvorgangs gibt.“
Offenbar brauchen mRNAs, auf denen eine hohe Anzahl an Ribosomen Platz findet, eine längere Zeit, bis sie ihre volle Kapazität erreicht haben. Kurzlebige mRNAs hingegen erreichen schneller ihre volle Produktivität, indem sie besonders schnell beladen werden. Wie aber Halbwertszeit und Beladungsgeschwindigkeit genau miteinander gekoppelt sind, bleibt noch aufzuklären.
Die Dauer des Beladungsvorgangs ist vor allem dann wichtig, wenn die Synthese von mRNAs sich schnell ändert, etwa wenn ein Makrophage, eine Fresszelle des Immunsystems, auf Bestandteile von Bakterien stößt und in kürzester Zeit eine Entzündungsreaktion in Gang setzt. Die Synthese zahlreicher mRNAs wird dabei herauf- und heruntergefahren. Wie schnell diese Änderung der mRNA-Spiegel letztendlich auch zu einer Änderung der Proteinproduktion führt, hängt im Wesentlichen von der Beladungsgeschwindigkeit der einzelnen mRNAs ab. Diese Ergebnisse konnte das Team gemeinsam mit ihren Partnern an der Tel Aviv University in Israel direkt auf Ebene der produzierten Proteine bestätigen.
Die Translationskontrolle spezifischer mRNAs spielt eine Schlüsselrolle bei der Regulierung der Genexpression. Die Ergebnisse der Mannheimer Forscher weisen die Ribosomenbeladung nun als einen wichtigen Teilaspekt der Translationskontrolle aus.
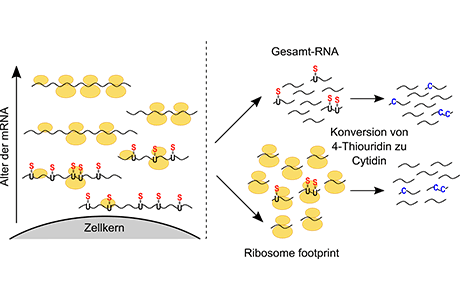
Messung der Ribosomenbeladung junger mRNAs
Um die Ribosomenbeladung junger mRNAs zu bestimmen, müssen die Forscher zunächst junge von älteren mRNAs unterscheiden können. Dazu markierten sie die mRNAs während ihrer Entstehung mit einem modifizierten Nukleotid (4-Thiouridin anstelle von Uridin). Die Beladung von mRNAs mit Ribosomen wurde mittels Ribosomen-Footprinting gemessen, wofür Ribosomen-beladene mRNA-Abschnitte isoliert und sequenziert werden. Der Anteil junger mRNAs an der gesamten translatierten mRNA wurde dabei bestimmt, indem 4-Thiouridin chemisch in das Nukleotid Cytidin umgewandelt wurde, was zu „Fehlern“ in der Sequenz der markierten mRNA-Fragmente führt. Auf diese Weise können Sequenzen „junger“ mRNAs bioinformatisch identifiziert werden (siehe Abbildung).
*Publikation
Johanna Schott, Sonja Reitter, Doris Lindner, Jan Grosser, Marius Bruer, Anjana Shenoy, Tamar Geiger, Arthur Mathes, Gergana Dobreva & Georg Stoecklin
Nascent Ribo-Seq measures ribosomal loading time and reveals kinetic impact on ribosome density
Nature Methods, 2021 Sep 3.
DOI: 10.1038/s41592-021-01250-z