Biochemiker am Institute for Innate Immunoscience (MI3) der Medizinischen Fakultät Mannheim der Universität Heidelberg haben ein Protein identifiziert, das als Kofaktor des mRNA-abbauenden CCR4-NOT Komplexes den globalen Abbau von Boten-RNA (mRNA) steuert. Diesem Kofaktor, RNF219 genannt, kommt damit eine wichtige Funktion in der Regulation der Genexpression zu.
mRNAs spielen eine zentrale Rolle bei der Produktion von Eiweißen (Proteinen) im Körper, deren Bauanleitungen in der Erbinformation (DNA) verschlüsselt im Zellkern vorliegen. mRNAs sind Abschriften (Transkripte) einzelner Gene, die jeweils die Anleitung für den Bau eines Proteins in sich tragen. Funktion der mRNA-Transkripte ist es, den Bauplan der körpereigenen Proteine aus dem Zellkern in das Zellplasma zu transportieren und dort in den Proteinfabriken (Ribosomen) der Zelle als Matrize für die Herstellung der Proteine zu dienen.
Die Genexpression, die darüber bestimmt, welche Proteine wann und in welcher Menge in der Zelle hergestellt werden, hängt stark von der Menge an mRNA ab, welche durch das Verhältnis von mRNA-Synthese und -Abbau reguliert wird. Beide Parameter spielen damit eine wichtige Rolle in der Regulation der Proteinbiosynthese.
Neben der sogenannten Cap-Struktur am Anfang einer mRNA bildet der Polyadenosin-Schwanz am Ende ein zentrales, stabilitätsbestimmendes Element fast aller mRNAs. In Eukaryoten – Organismen, deren Erbinformation in einem Zellkern verpackt ist – stellt die Verkürzung der Polyadenosin-Schwänze (Deadenylierung) meist den einleitenden und oft auch geschwindigkeitsbestimmenden Schritt des mRNA-Abbaus dar. Vermittelt wird dieser Prozess hauptsächlich durch den evolutionär konservierten CCR4-NOT* Komplex im Zellplasma.
CCR4-NOT ist ein Multi-Proteinkomplex, der sich aus mehreren Untereinheiten zusammensetzt und viele Aspekte des mRNA Metabolismus reguliert. Dazu gehören die Transkription sowie die von der Deadenylierung abhängige Translation und der mRNA-Abbau.
In einer früheren Studie hatte das Forscherteam um Georg Stoecklin bereits gezeigt, dass der globale mRNA-Abbau maßgeblich durch die Acetylierung des CCR4-NOT Komplexes gefördert wird, etwa wenn Zellen mit sogenannten Histon-Deacetylase-Hemmern behandelt werden**.
In ihrer aktuellen Studie untersuchten die Forscher, ob Acetylierung zu einer Änderung in der Zusammensetzung des CCR4-NOT Komplexes führt. Durch CRISPR/Cas9-vermittelte Proteinmarkierung konnte Fabian Poetz den zelleigenen CCR4-NOT Komplex aufreinigen und das Protein RNF219 als einen Acetylierungs-abhängigen Kofaktor des Komplexes identifizieren.
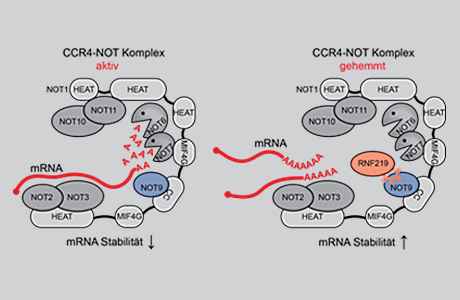
Die Ubiquitin-Ligase RNF219 hemmt den globalen mRNA Abbau
Das Forscherteam konnte daraufhin zeigen, dass RNF219 eine aktive E3 Ligase ist, die ein kleines Regulatorprotein, Ubiquitin genannt, auf andere Proteine übertragen kann. Durch eine genomweite Messung von mRNA Halbwertszeiten konnten sie nachweisen, dass RNF219 in den Zellen den Abbau einer sehr großen Anzahl von mRNAs hemmt. Die durch Acetylierung induzierte Verminderung der RNF219 Expression trägt also zur Beschleunigung des globalen mRNA Abbaus bei, wie sie beispielsweise nach der Behandlung mit Histon-Deacetylase-Hemmern beobachtet wird.
Weitergehende biochemische Untersuchungen in Zusammenarbeit mit einem Team am National Cancer Institute in den USA zeigten, dass RNF219 direkt an die NOT9 Untereinheit des CCR4-NOT Komplexes bindet und dessen Deadenylierungs-Aktivität vermindert (Mechanismus in der Abbildung). Mittels im Computer simulierter (in silico-) Modellierung der Proteinstruktur und anschließender Validierung konservierter Interaktionsstellen konnte die genaue Bindung von RNF219 an NOT9 dargestellt werden.
Steuerung der Expression hoch reaktiver Gene
Die genomweite Messung von mRNA Halbwertszeiten zeigte zudem, dass instabile mRNAs sogenannter immediate early genes, einer Gruppe hoch reaktiver Gene, welche am schnellsten auf zelluläre Stimulation reagieren, in besonderem Maße von der RNF219-vermittelten Stabilisierung abhängig sind. Diese Experimente untermauern die Bedeutung der aktiven Regulation des mRNA Abbaus für die dynamische Expression solch hoch reaktiver Gene, deren mRNAs typischerweise sehr kurzlebig sind.
Noch ungeklärt ist die Frage, warum die Ubiquitin-Ligase Aktivität von RNF219 nur für die Stabilisierung langlebiger mRNAs nötig ist, hingegen kurzlebige mRNAs auch durch eine RNF219 Variante stabilisiert wurden, die keine Ubiquitin-Ligase Aktivität besitzt. Die genauen Zusammenhänge zwischen dem Ubiquitin-System und der Regulation der mRNA Stabilität sind noch weitgehend unerforscht, und werden vom Team um Georg Stoecklin in Zukunft weiterverfolgt.
Publikation
Fabian Poetz, Joshua Corbo, Yevgen Levdansky, Alexander Spiegelhalter, Doris Lindner, Vera Magg, Svetlana Lebedeva, Jörg Schweiggert, Johanna Schott, Eugene Valkov & Georg Stoecklin
RNF219 attenuates global mRNA decay through inhibition of CCR4-NOT complex-mediated deadenylation.
Nat Commun 12, 7175 (2021).
DOI: https://doi.org/10.1038/s41467-021-27471-6
* Carbon Catabolite Repression-Negative On TATA-less
** Sharma et al., Mol. Cell 2016